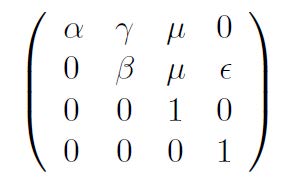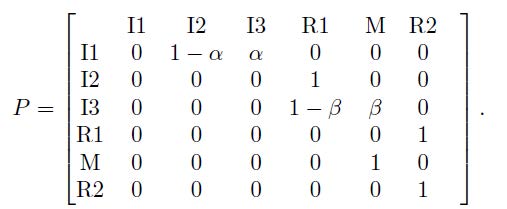Por Manuel de León y Antonio Gómez Corral.- Esta entrada fue publicada en el blog Matemáticas y sus fronteras , el día 21 de mayo de 2020.
Las cadenas de Markov se han revelado de una enorme utilidad en numerosos campos, en particular, en la predicción y el control del desarrollo de una epidemia. En esta entrada vamos a considerar dos situaciones diferentes, referidas al virus VIH del SIDA y al virus SARS-CoV-2 de la Covid-19, analizadas mediante dos modelos sencillos.
Andrey Markov
Como habíamos comentado en entradas anteriores, una cadena de Markov es un proceso estocástico o conjunto de variables aleatorias (en cantidad discreta o continua, según el contexto) caracterizadas por la propiedad Markoviana, es decir, se refleja que el valor o estado de la variable asociada a un instante concreto de tiempo determina el siguiente estado del sistema en estudio, pero éste no depende de los estados asociados a las variables aleatorias anteriores. En otras palabras, “el futuro depende del pasado, pero sólo a través del presente”.
Las aplicaciones de las cadenas de Markov en el estudio de epidemias se desarrollan en paralelo a aquéllas realizadas desde el uso de ecuaciones diferenciales. El lector habitual de este blog puede observar ese paralelismo en dos de nuestras recientes entradas. En concreto, el modelo SIR en la entrada Las matemáticas del coronavirus Covid-19 es construido en el contexto determinístico, mientras que nuestros comentarios en la entrada Las matemáticas contra la malaria y el modelo SIR destacaban el papel crucial de Anderson Grey McKendrick en la formulación estocástica del modelo SIR.
Es importante incidir sobre las diferencias fundamentales entre el mundo determinista y el mundo estocástico, tanto para identificar la herramienta matemática usada como para entender el objeto en estudio. A pesar de que hablamos en ambos casos de ecuaciones diferenciales, éstas son concebidas de manera diferente:
- En el contexto determinista, las ecuaciones diferenciales regulan la evolución de los números de susceptibles, de infectados y de recuperados – equivalentemente, sus proporciones – en cada instante de tiempo t, concibiendo estos números como funciones de t (el tiempo), de modo que cada uno de estos números es una función real de variable real.
- En el contexto estocástico, las ecuaciones diferenciales involucran a la distribución de probabilidad conjunta de los números de susceptibles, de infectados y de recuperados en el instante de tiempo t, concibiendo estos números como variables aleatorias y tenemos, en el caso del modelo SIR, tres variables aleatorias asociadas (los números de susceptibles, infectados y recuperados) a cada instante de tiempo t.
Como consecuencia de lo anterior, la solución del sistema de ecuaciones diferenciales en un contexto determinista permite dibujar los números de susceptibles, de infectados y de recuperados en función del tiempo y la curva representando a uno de estos números, por ejemplo, el número de infectados I(t) en el instante t, sobre unos ejes cartesianos es única como función real de t. Por el contrario, cuando se resuelve el sistema de ecuaciones diferenciales en el contexto estocástico se obtiene la distribución de probabilidad conjunta de los números de susceptibles, de infectados y de recuperados en cada instante de tiempo t. Si, por ejemplo, nos centramos en el número de infectados I(t) en el instante t, entonces es posible dibujar la probabilidad de que I(t) = i como una curva como función de t, para cada valor i entre 0, 1, …, N, siendo N el tamaño total de la población. Esas curvas, y tenemos una para cada valor i, expresan cómo de verosímiles son cada uno de los valores posibles de la variable aleatoria I(t). Informalmente hablando, esto equivale a lo siguiente:
Si fuéramos capaces de reproducir un número n grande de situaciones prácticas de la propagación de una epidemia de tipo SIR entre los individuos de una población y contabilizásemos, en un instante de tiempo t concreto, la frecuencia relativa de aparición de cada valor i entre esas n situaciones, esas frecuencias relativas serían una estimación de la probabilidad de que I(t) = i, tanto más precisa cuanto mayor sea el número grande n de situaciones observadas.
Andrey Kolmogorov
En este punto, la llave que formaliza el comportamiento asintótico que, cuando n tiende hacia infinito, subyace en nuestro comentario es uno de los resultados fundamentales de la Teoría de la Probabilidad moderna, las leyes de los grandes números, que permiten acceder a la definición axiomática de probabilidad – formulada por el matemático ruso Andrey Nikolaevich Kolmogorov (Tambov, 1903 – Moscú, 1987) – desde la noción de probabilidad frecuentista vigente hasta principios de 1930. Para ello es necesario aludir a la convergencia en probabilidad (ley débil) o a la convergencia casi segura (ley fuerte).
Una vez hecha, quizás no muy brevemente, esta puntualización sobre modelos deterministas versus modelos estocásticos, nos centramos en el contexto estocástico y comentamos sobre dos modelos de epidemias basados en cadenas de Markov. Hemos usado dos artículos científicos en los que el lector interesado en profundizar más allá de este blog podrá encontrar más detalles y bibliografía.
La epidemia del VIH/SIDA
En el artículo titulado “Modelo estocástico para la epidemia del VIH/SIDA”, de Erick Manuel Delgado-Moya y Aymée Marrero-Severo, se construye una sencilla cadena de Markov de la siguiente forma:
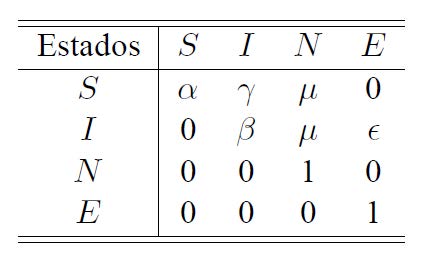
Se definen, como en las variantes del modelo SIR, cuatro posibles estados para un individuo de la población en cuestión: S (susceptible), I (infectado), N (individuo muerto por muerte natural) y E (individuo muerto a causa de la enfermedad). Como unidad de tiempo se emplea el año, generando entonces una cadena de Markov en tiempo discreto cuando el estado de un individuo es registrado, por ejemplo, el día 1 de marzo de cada año, si éste se sometiera a pruebas diagnósticas con una periodicidad anual. Las probabilidades asociadas a la evolución del estado de un individuo entre dos etapas consecutivas, es decir, desde una revisión diagnóstica y la siguiente, se denotan por
α: probabilidad de mantenerse en el estado de susceptible.
β: probabilidad de mantenerse en el estado de infectado.
μ: probabilidad de muerte natural.
γ: probabilidad de, estando en el estado susceptible, pasar al estado de infectado.
ε: probabilidad de muerte por la enfermedad, dado que está en el estado infectado.
Debemos recordar que, para obtener una matriz de transición, se deben cumplir las relaciones
α+γ+μ= 1 y β+μ+ ε = 1.
Las probabilidades anteriores están ligadas a las transiciones
de modo que la matriz de transición viene dada por
Sin entrar en los detalles del estudio de Erick Manuel Delgado-Moya y Aymée Marrero-Severo, una cuestión interesante está asociada a cómo influir para mejorar los resultados en el tratamiento terapéutico de un paciente. Tendríamos, por tanto, que influir sobre el parámetro ε, que se suele parametrizar como una función o índice de eficacia. Claro que, si reducimos la mortalidad con el tratamiento, también podríamos estar contribuyendo a aumentar el número de infectados.
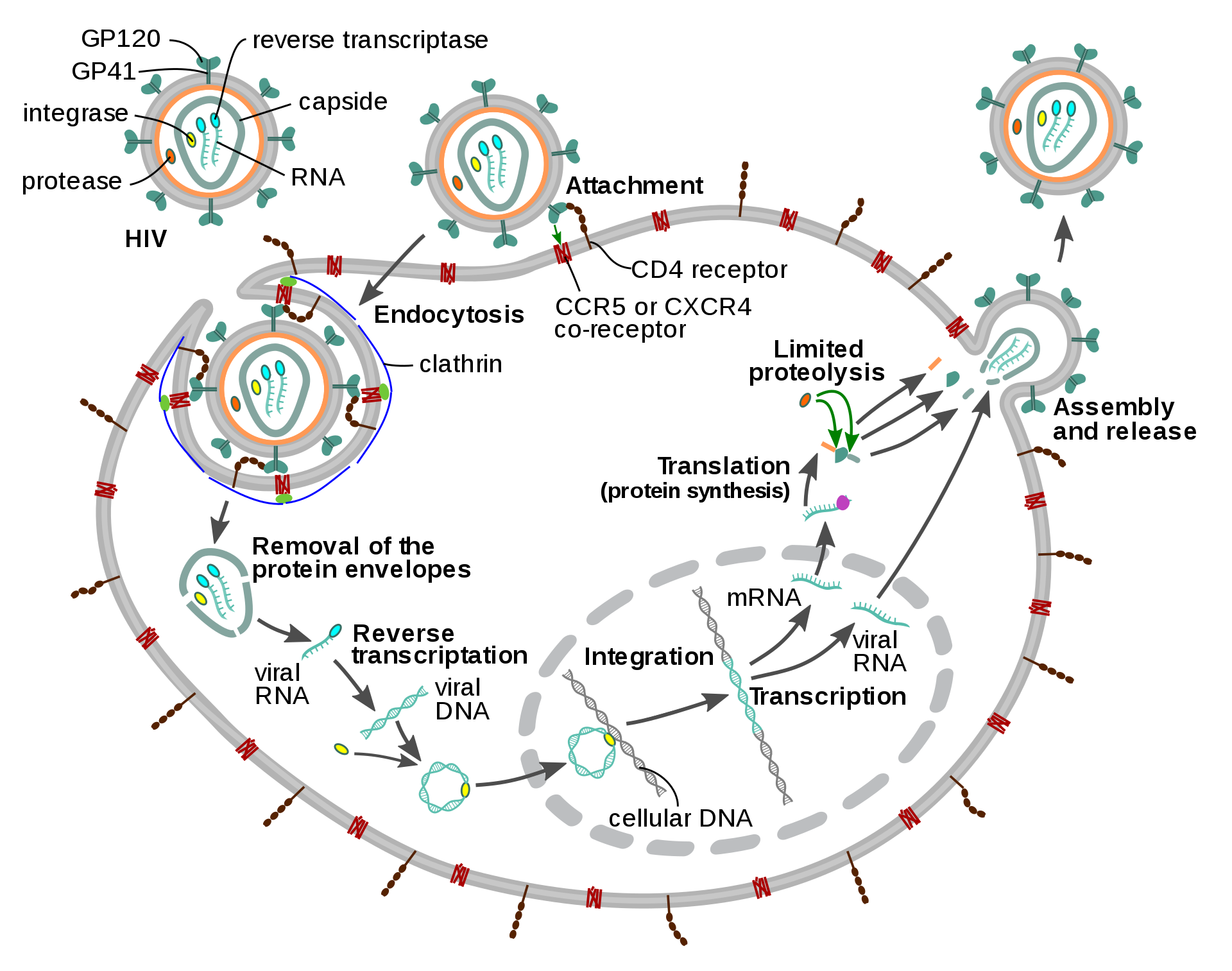
Ciclo de replicación del virus del SIDA
Surge entonces un interesante problema vinculado al control del número de infectados que, en el caso del SIDA, se puede abordar desde la abstinencia, la reducción de las prácticas sexuales o el uso de barreras profilácticas (preservativos), así como desde los beneficios de un adecuado tratamiento terapéutico. Como un procedimiento alternativo al realizado por los autores, mencionamos que es posible abordar el problema de control – resultante desde la introducción de costes – usando la teoría de la decisión Markoviana que combina cadenas de Markov con técnicas de Optimización Matemática.
La lucha contra el coronavirus SARS-CoV-2 de la Covid-19
Describimos ahora, de manera concisa, el modelo diseñado en el reciente artículo “COVID-19: Estimating spread in Spain solving an inverse problem with a probabilistic model”, de Marcos Matabuena, Carlos Meijide-García, Pablo Rodríguez-Mier y Víctor Leborán. Con nuestros comentarios no pretendemos reproducir su contenido, sino poner de manifiesto cómo es posible partir de un modelo sencillo, basado en una cadena de Markov, y generar una variante más avanzada prescindiendo de los principios Markovianos.
Previamente, es obligado hacer dos observaciones necesarias para entender cómo describir una cadena de Markov en tiempo continuo desde elementos más sencillos. En concreto, una cadena de Markov en tiempo continuo, denotada por {X(t): t≥0}, podemos describirla a través de combinar dos elementos:
- La sucesión de estados visitados, {Xn: n=0,1,…}, que resulta ser una cadena de Markov en tiempo discreto, denominada cadena encajada, y tiene una matriz de transición específica.
- La sucesión de tiempos de permanencia en los estados visitados que, en el supuesto de que el proceso acceda al estado i, implica que el tiempo de permanencia en ese estado es una variable aleatoria exponencial de parámetro α(i), de manera que el tiempo medio de permanencia en el estado i es 1/α(i), y no depende de los estados anteriormente visitados antes de acceder a i, ni del estado que se visitará cuando el proceso abandone i.
El lector avanzado dentro de la teoría de procesos estocásticos observará que la anterior descripción se refiere a una cadena de Markov en tiempo continuo regular y no de cualquiera, pero esta descripción es suficiente para nuestro objetivo en este ejemplo.
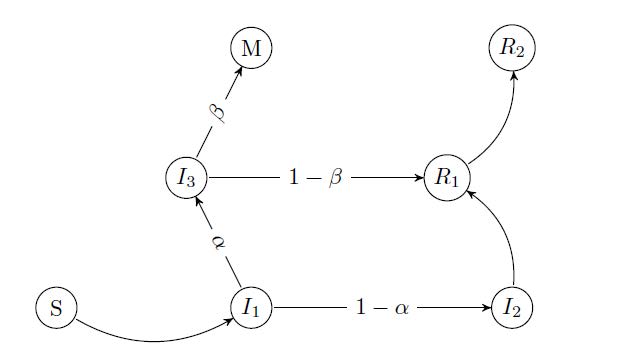
En este caso, el modelo de Marcos Matabuena, Carlos Meijide-García, Pablo Rodríguez-Mier y Víctor Leborán contiene las siguientes variables:
S(t): número de individuos susceptibles en el instante t
I1(t): número de individuos infectados que están incubando el virus en el instante t
I2(t): número de individuos infectados que han pasado el periodo de incubación, pero no muestran síntomas de la enfermedad en el instante t
I3(t): número de individuos infectados que han pasado el periodo de incubación y presentan síntomas en el instante t
R1(t): número de individuos recuperados que son todavía capaces de infectar a otros en el instante t
R2(t): número de individuos recuperados que no son capaces de infectar a otros en el instante t
M(t): número de fallecidos acumulados hasta el instante t
Entonces, I(t) = I1(t) + I2(t) +I3(t) representa el número total de infectados en el instante de tiempo t, y R(t) = R1(t) + R2(t) es el número de recuperados.
El siguiente gráfico es una representación esquemática de las transiciones entre los siete compartimentos o subpoblaciones que las variables anteriores generan:
Representación esquemática tomada desde el artículo de Marcos Matabuena, Carlos Meijide-García, Pablo Rodríguez-Mier y Víctor Leborán, arxiv.org/abs/2004.13695
Si queremos alimentar la sopa de letras surgida desde el modelo SIR, entonces la anterior figura es un ejemplo de otra variante del modelo SIR.
El modelo construido para las anteriores variables aleatorias sirve a los autores para extraer interesantes conclusiones para España, con comentarios específicos sobre sus Comunidades Autónomas, aunque no lo hagan directamente desde una cadena de Markov en tiempo continuo, sino un proceso inspirado en ella. En concreto, usan una matriz de transición de la cadena encajada de la forma
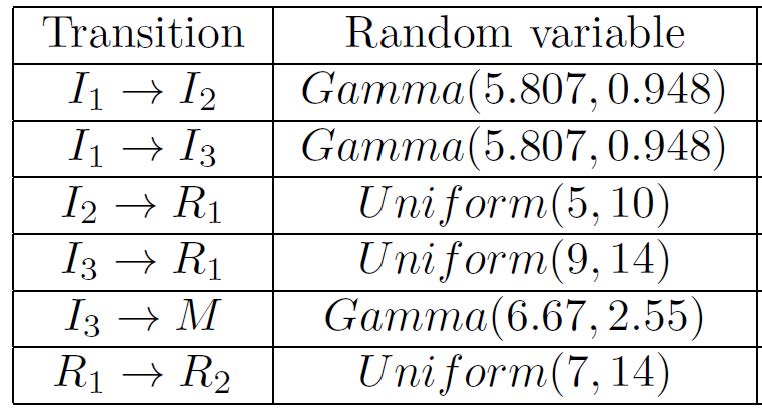
Sin embargo, la experiencia de los autores y la literatura existente ha llevado a éstos a reemplazar la hipótesis de exponencialidad sobre los tiempos de permanencia por otras distribuciones de probabilidad que hacen que el proceso estocástico (S(t), I1(t), I2(t), I3(t), R1(t), R2(t), M(t)) no sea Markoviano, pero sí se ajuste mejor a la incidencia del virus SARS-CoV-2 sobre la población española. Aparentemente, la dependencia de los estados de destino en las transiciones en la siguiente tabla (por ejemplo, el tiempo de permanencia en el estado I3 depende del estado final de transición R1 y M) nos lleva a sospechar que el proceso (S(t), I1(t), I2(t), I3(t), R1(t), R2(t), M(t)) no conserva, ni tan siquiera, las virtudes de un proceso de Markov determinista por partes (piecewise-deterministic Markov process). El modelo resultante no es estacionario y tiene una estructura de dependencia compleja.
Las distribuciones usadas por Marcos Matabuena, Carlos Meijide-García, Pablo Rodríguez-Mier y Víctor Leborán.
Para el resto de detalles, de tanta actualidad en los días que vivimos, remitimos al lector al artículo original de los autores.